Превосходит зарубежные аналоги: новый российский алгоритм исследования белков позволит изучить эволюционные изменения, произошедшие сотни миллионов лет назад
Биоинформатики ФИЦ Биотехнологии РАН доказали, что созданная ими математическая модель для сопоставления последовательностей у белков, разошедшихся на эволюционном древе сотни миллионов лет назад, превосходит лучшие зарубежные аналоги, сообщила пресс-служба ФИЦ Биотехнологии РАН. Результаты исследования на масштабной выборке в 490 белковых семейств опубликованы в журнале Symmetry.
Белки – это незаменимые для живых клеток молекулы со множеством различных функций. Именно их строение и многообразие «записано» в нашей ДНК. Каждый белок – это длинная цепь, состоящая из звеньев-аминокислот, которые могут иметь различную форму. Сравнивая их последовательность у «родственных» белков, ученые находят сохраняющиеся мотивы – это короткие похожие отрывки, связанные с выполнением одной биологической функции. Благодаря их изучению можно узнавать больше о работе белков и строить эволюционные деревья организмов. Поэтому поиск мотивов – одна из важнейших задач вычислительной биологии.
Для него часто используется множественное выравнивание, при котором «родственные» последовательности располагают друг над другом и сравнивают при помощи математических моделей. Одна из таких моделей легла в основу предсказывающего пространственную структуру белков алгоритма AlphaFold, разработчики которого получили Нобелевскую премию по химии в 2024 году. Однако если аминокислотные последовательности сильно отличаются, существующие методы могут оказаться неточными, пропуская мотивы или находя их там, где их на самом деле нет. Исследователи ФИЦ Биотехнологии РАН создали собственный алгоритм для множественного выравнивания, MADHS, который снижает частоту подобных ошибок. В новой работе они проверили эффективность метода на масштабной выборке последовательностей семейств белков, которые за свою историю накопили много отличий.
«Это уникальная работа, которая демонстрирует, что MADHS выравнивает аминокислотные последовательности лучше всех известных на сегодняшний день программ. Мы подтвердили этот результат для 490 известных белковых семейств – это все известные белковые семейства с подобием ниже 20%», – рассказал соавтор статьи профессор Евгений Коротков, руководитель группы математического анализа последовательностей ДНК и белков ФИЦ Биотехнологии РАН.
Аббревиатура MAHDS расшифровывается как multiple alignments of highly diverged sequences, или метод множественного выравнивания высоко дивергентных (то есть, накопивших много отличий и сильно разошедшихся в процессе эволюции) последовательностей. Созданный российскими биоинформатиками в 2021 году, он показал свои преимущества по сравнению с аналогами – T-Coffee, MUSCLE, Clustal Omega, Kalign, MAFFT и PRANK.
Проверить статистическую значимость результата исследования биоинформатикам помогла группа методов Монте-Карло. В основе этого подхода – математическая модель процесса, которая позволяет просчитать вероятность различных результатов при помощи генерирования случайных чисел. Согласно выводам ученых, MAHDS стал самым подходящим методом анализа последовательностей, которые принадлежат разошедшимся сотни миллионов лет назад эволюционным группам.
«Мы показали, что метод MAHDS позволяет получить статистически значимые результаты там, где остальные методы выдают совершенно случайный вариант множественного выравнивания последовательностей. Наш новый математический метод уже работает, и мы создали доступный для любого пользователя сервер», – рассказал соавтор публикации Дмитрий Костенко, младший научный сотрудник группы математического анализа последовательностей ДНК и белков ФИЦ Биотехнологии РАН.
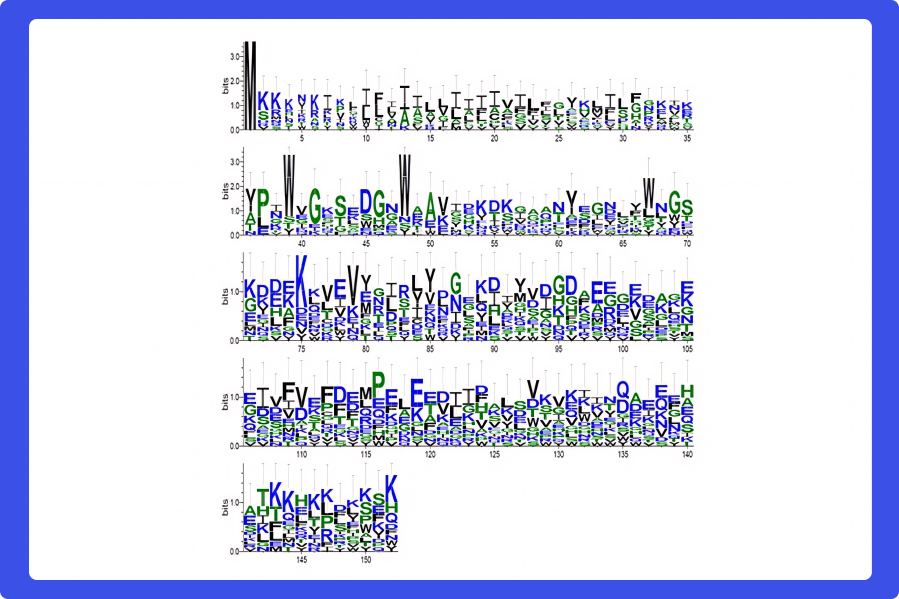
На рисунке показан вклад каждой аминокислоты во множественное выравнивание белков из семейства PF16302. Видно, что большинство столбцов содержат несколько доминирующих аминокислот, что указывает на статистически значимое выравнивание, несмотря на слабое сходство аминокислотных последовательностей, входящих в семейство. Источник: ФИЦ Биотехнологии РАН.